Новая техника печати на ДНК может произвести революцию в хранении данных

Новая техника кодирования позволяет «печатать» и хранить данные на ДНК подобно старомодному печатному станку. Эпигенетическая система маркировки используется для записи данных в двоичном коде, а нанопоровое устройство секвенирования — для считывания результатов. Несмотря на кажущуюся сложность конструкции, этот метод не требует особых знаний в области лабораторных технологий.
Несмотря на современные достижения в области хранения данных, они еще далеки от того, чтобы сравниться с ДНК. Концентрируя огромные объемы информации в крошечных объемах, один кубический сантиметр ДНК может хранить эквивалент 10 эксабайт (ЭБ) данных в течение тысяч лет или даже миллионов при наилучших условиях. Благодаря такому огромному потенциалу ДНК в настоящее время изучается как новый носитель для хранения цифровой информации.
Современные технологии предполагают преобразование данных в двоичном коде в последовательности нуклеотидов (аденин, гуанин, цитозин и тимин). Запись осуществляется путем синтеза нити ДНК с нуля, то есть путем добавления нуклеотидных оснований одного за другим в заранее определенном порядке. Однако эти методы ограничены с точки зрения скорости записи и длины синтезируемой нити.
Чтобы преодолеть эти ограничения, были предложены альтернативные подходы, такие как параллельная запись. Однако структурная нестабильность и ограниченная производительность обработки информации препятствуют их применению. В отличие от этого, эпигеном может кодировать модифицируемую информацию, которая стабильнее геномной последовательности, несущей ее. В исследовании, проведенном под руководством Пекинского университета, предложена методика, вдохновленная древними техниками печати и основанная на передаче эпигеномных признаков.
«Вдохновленные естественным эпигеномным наследованием и синтетической самосборкой ДНК, мы разработали нетрадиционную систему записи данных на ДНК, которая позволяет параллельно записывать произвольные биты эпигенетической информации (эпи-биты) на шаблоны ДНК на основе ферментативного метилирования под руководством самосборки ДНК», — объясняют исследователи в работе, опубликованной в журнале .
Система, вдохновленная старыми печатными станками
Методика предполагает кодирование бинарных данных непосредственно на уровне ДНК с помощью селективного метилирования — эпигенетической модификации. Если основание метилировано, оно кодируется как 1, а если не метилировано, то как 0. Другими словами, «информацию несет не сама последовательность [ДНК], а слой метилирования», — объясняет соавтор исследования Лонг Цянь из Пекинского университета в журнале Chemical & Engineering News.
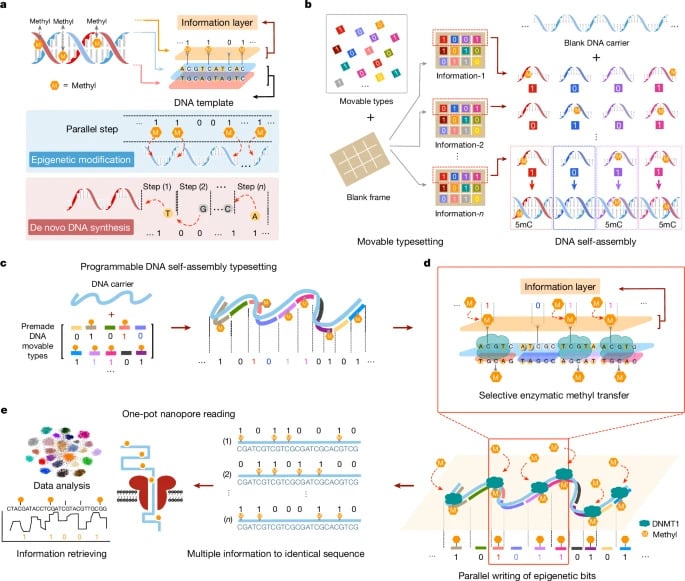
Исследователи разработали программируемый метод письма с помощью самосборки, похожий на подвижный шрифт, использовавшийся в старых типографиях. Для печати газет их собирали по одному на больших пластинах, чтобы сформировать текст для печати, а затем наносили на них чернила, после чего по ним проходила бумага. В технике, использованной в исследовании, в качестве бумаги выступает универсальный шаблон одноцепочечной ДНК, а печатный шрифт представляет собой набор из 700 коротких нитей, называемых «кирпичиками». Каждый кирпичик состоит из 24 нуклеотидов, а цитозины являются местами метилирования.
Чтобы закодировать информацию, кирпичики располагаются в шаблоне ДНК в определенной последовательности, каждый из которых содержит эпибит информации. ДНК обрабатывается метилтрансферазой (ферментом), которая распознает метилированный цитозин и впечатывает информацию о метилировании, связываясь с шаблоном ДНК. Этот процесс можно сравнить с тем, как цифровые данные копируются на жесткие диски путем изменения направления намагничивания. Затем информация восстанавливается с помощью метода нанопорового секвенирования, позволяющего непосредственно в реальном времени считывать фрагменты ДНК и состояние их метилирования.
Команда протестировала свою технику печати ДНК для кодирования рисунка тигра и фотографии панды, представляющих собой 16 833 и 252 500 бит соответственно. Все данные были успешно напечатаны и восстановлены. В то время как предыдущие методы требовали от 4 до 20 минут для добавления нуклеотидов один за другим, этот прогресс позволяет одновременно печатать 350 бит информации намного быстрее, чем нынешние методы.

Доступность для неспециалистов и точность 98,58 %
Чтобы оценить доступность методики, исследователи пригласили 60 добровольцев, не имеющих опыта работы с лабораторными методами, воспользоваться платформой для резервного копирования и восстановления данных. Участникам было предложено использовать программное обеспечение под названием iDNAdrive для кодирования текстов по своему выбору общим объемом около 5 000 бит. В то время как хранение нуклеотидов должно осуществляться на специализированной платформе для синтеза и требует развитых лабораторных навыков, манипуляции, необходимые для этой новой техники, сводятся к добавлению различных реагентов в одну пробирку. Данные были распечатаны и считаны с точностью 98,58%.
Исследователи планируют и дальше совершенствовать методику, уделяя особое внимание увеличению скорости записи данных и снижению затрат. Они также надеются увеличить объем памяти, распространив эпигенетическую маркировку на другие нуклеотидные основания, а не ограничиваясь только цитозином. В конечном счете, эта технология может произвести революцию в области хранения данных, не в последнюю очередь благодаря своей доступности.